|
|
|
|
|

| Participants |
Contexte Scientifique |
Objectifs |
Résultats du projet | Animation | Publications |
Participants
|
IRISA / INRIA Rennes - Campus Universitaire de Beaulieu, Avenue du Général Leclerc, 35042 Rennes Cedex - France |
| Charles Kervrann, Chargé de Recherche INRA (Coordinateur du Projet) Patrick Bouthemy, Directeur de Recherche INRIA Jérome Boulanger, Doctorant |
|
INRA - Unité MIA de Jouy-en-Josas, Centre de Recherche de Jouy-en-Josas, Domaine de Vilvert, 78352 - Jouy-en-Josas - France |
| Alain Trubuil, Ingénieur de Recherche INRA (Responsable équipe 1) Kiên Kiêu, Chargé de Recherche INRA Ikhlef Bechar, Doctorant |
|
Institut Curie - UMR 144 - CNRS - 26, rue d'Ulm, 75248 Paris Cedex 05 - France |
| Jean-Baptiste Sibarita, Ingénieur de Recherche CNRS (Responsable équipe 2) Jean Salamero, Chargé de Recherche CNRS Victor Racine, Doctorant |
|
Interactions Cellulaires et Moléculaires - UMR 6026, Université de Rennes 1, Campus Universitaire de Beaulieu, Bâtiment 13, 35042 Rennes Cedex - France |
| Denis Chrétien, Chargé de Recherche CNRS (Responsable équipe 3) Isabelle Arnal, Chargé de Recherche CNRS |
Contexte Scientifique
Le développement de la biologie
intégrative se caractérise par la mise en oeuvre de nouvelles techniques qui
produisent des données en masse et de nature variée. Seules l'analyse et
l'interprétation réalisées automatiquement permettront aux chercheurs d'y faire
face ; tous les domaines sont concernés: analyse des séquences, des données
d'expression, des données-images, des données bibliographiques, ... Dans ce projet, notre
objectif est de faciliter l'extraction et la structuration automatiques des informations
dynamiques contenues dans des séquences temporelles d'images obtenues en
vidéo-microscopie nD+t. L'imagerie dynamique doit aider notamment à
comprendre la fonction de certaines protéines d'intérêt.
Les séquences temporelles d'images dont nous disposons se singularisent par un nombre
élevé de dimensions (deux ou trois dimensions spatiales et une dimension temporelle
pour autant de canaux spectraux) si on se réfère aux séquences vidéo (deux dimensions spatiales et une dimension temporelle).
Si plusieurs composantes spectrales
sont observées, on parle davantage d'imagerie 5D.
La vidéo-microscopie se distingue aussi de l'imagerie médicale plus répandue
(échographie, IRM, ...) par la nature des objets observés (organites de la cellule),
par le nombre élevé d'éléments mobiles de petite taille assimilables à des
particules (chromosomes, vésicules, ...), par la
complexité des dynamiques mettant en jeu plusieurs entités ou groupes d'entités
en interaction, par les phénomènes particuliers de coalescence des entités surtout
liés à des problèmes de résolution d'image.
Le corpus de données considéré pour l'analyse comparative
d'une expérience comportant plusieurs acquisitions est donc particulièrement
massif (plusieurs dizaines de Go) et particulièrement difficile à interpréter
qualitativement et quantitativement.
Faciliter la structuration et l'interprétation de l'information associées à de
tels corpus est désormais une nécessité pour de nombreux laboratoires de
biologie ; il est en effet devenu de plus en plus évident que la localisation
et la conformation spatio-temporelle de nombreuses constructions de la cellule est un
élément clé pour la compréhension de mécanismes fonctionnels. Cette problématique représente aussi un véritable défi scientifique en mathématiques appliquées et en informatique.Objectifs
La vidéo-microscopie visée dans ce projet, se singularise par la nature des objets observés (organites de la cellule), par le nombre souvent élevé d'éléments mobiles de petite taille, par la complexité des dynamiques mettant en jeu plusieurs entités ou groupes d'entités en interaction, par les phénomènes particuliers de coaslescence des entités surtout liés à des problèmes de résolution d'images. D'une manière générale, les modèles explicites et analytiques de mouvement en imagerie et vidéo sont mal adaptés pour traiter ces situations dynamiques complexes. Pour toutes ces raisons, des développements nouveaux s'appuyant notamment sur des techniques d'apprentissage, de modélisation stochastique (processus markoviens), d'analyse de mouvement apparent dans des séquences d'images et des techniques de suivi temporel (filtrage particulaire) des éléments dynamiques caractérisant les entités biologiques sous-jacentes, sont envisagées. Dans ce projet. il s'agit d'aborder les deux problématiques suivantes, insuffisamment traitées dans la littérature:- Construction de descripteurs de mouvement bas-niveaux en vidéo-microscopie (trajectoires nD+t, ...) ;
- Analyse et apprentissage statistiques des dynamiques nD+t.
Pour cela nous disposons de séquences temporelles d'images 3D acquises par microscopie champ-large a déconvolution rapide mise au point à l'Institut Curie (UMR 144) dont les caractéristiques sont les suivantes:
- Temps d'exposition: 30 msec / plan 2D
- Taille de chaque image 3D : 512 x 512 x 10 voxels
- Taille du voxel: 160 nm x 160 nm x 300 nm
- Fréquence temporelle: 3 volume / sec
- Taille de la séquence: 300 volumes (800 Moctets)
Résultats du projet
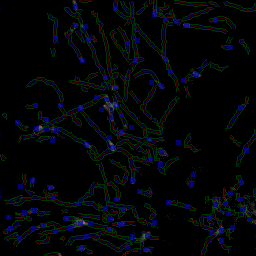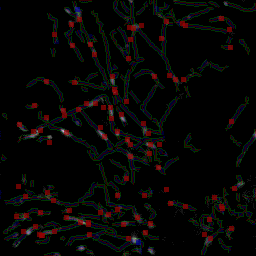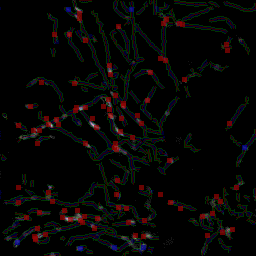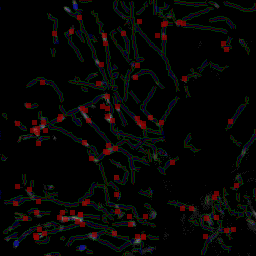
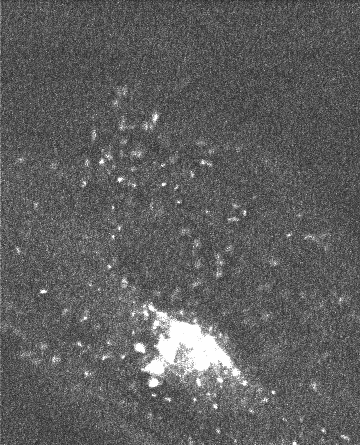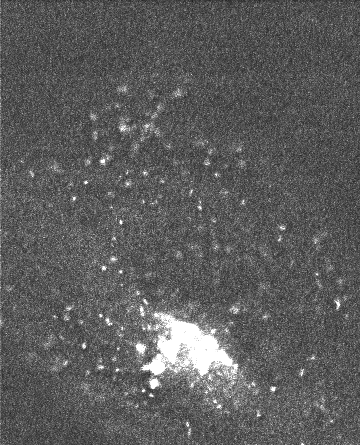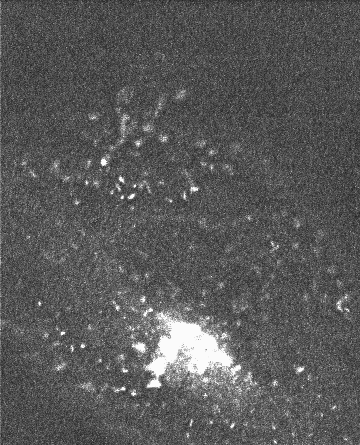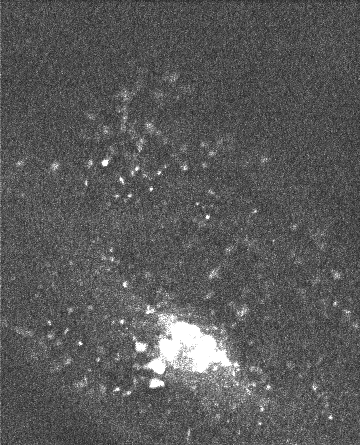
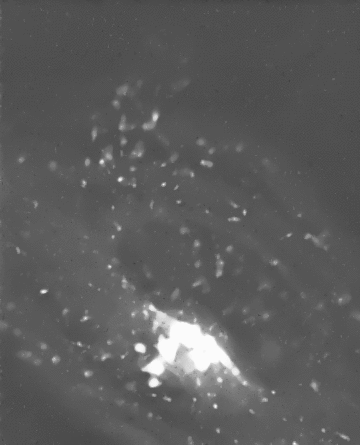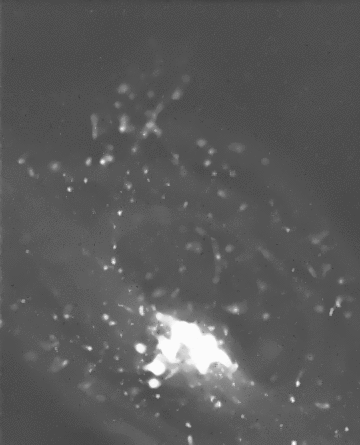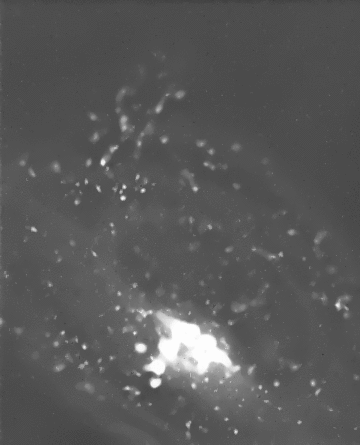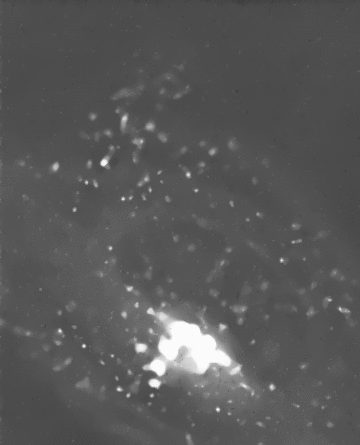
Débruitage spatio-temporel 2D/3D d'une séquence de vidéo-microscopie Rab6A'
|
J. Boulanger, C. Kervrann, P. Bouthemy. An adaptive statistical method for 4d-fluorescence image sequence
denoising with spatio-temporal discontinuities preserving . In Biophotonics for Life Sciences and Medicine , F. Faugel, P. Smigielski, A. Brandenburg, J.Fontaine, Eds., Fontis Media SA, Lausanne, Switzerland, pp. 97-113, 2006. Abstract. We present a spatio-temporal filtering method for significantly increasing the signal-to-noise ratio in noisy fluorescence microscopic image sequences where small particles have to be tracked from frame to frame. Image sequence restoration is achieved using a spatio-temporal adaptive window approach with an appropriate on-line window geometry specification. We have applied this method to noisy synthetic and real 4D images where a large number of small fluorescently labeled vesicles are moving in regions close to the Golgi apparatus. The SNR is shown to be drastically improved and the enhanced vesicles can be detected. This novel approach can be further used for biological studies where the dynamics of small objects of interest have to be analyzed in molecular and sub-cellular bio-imaging.
|
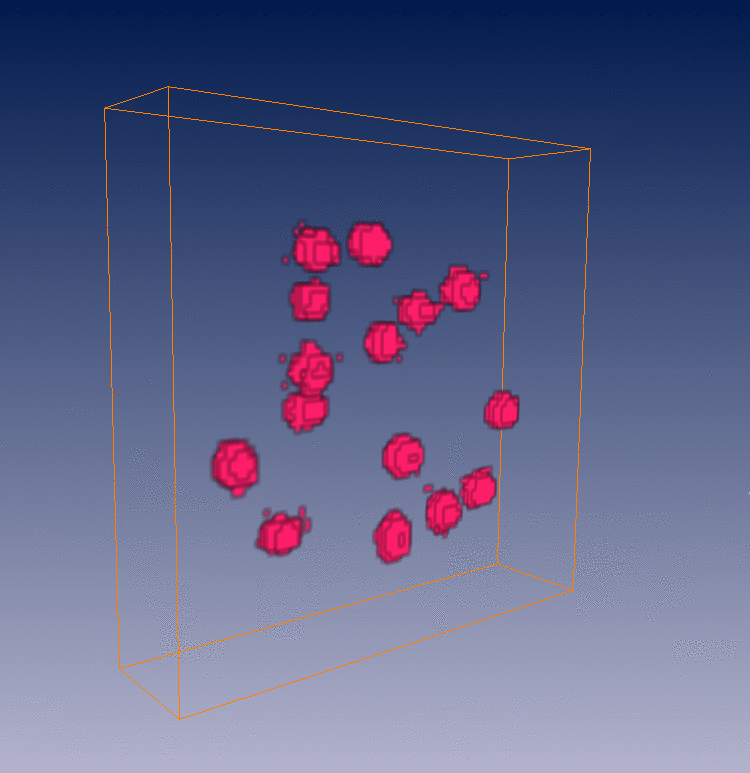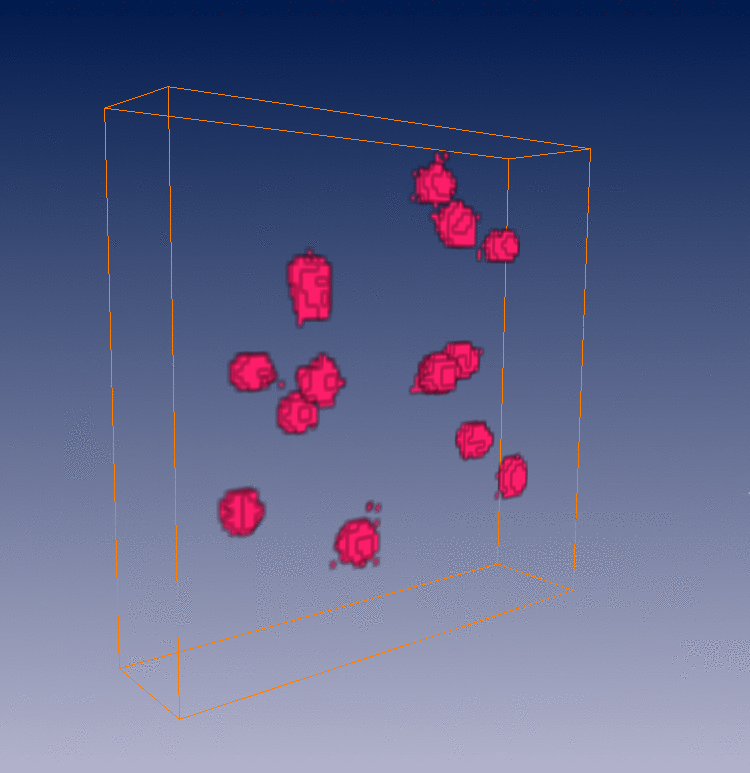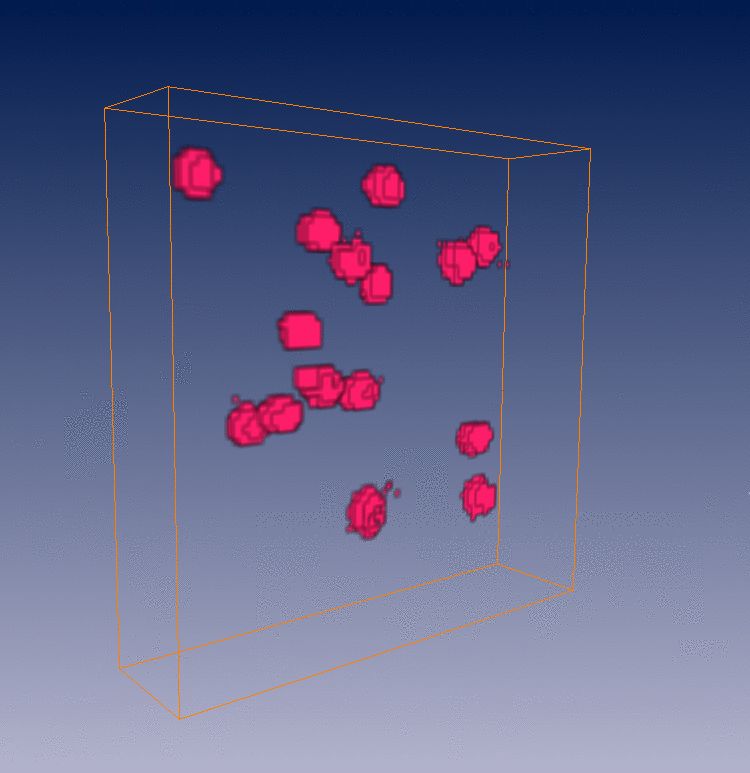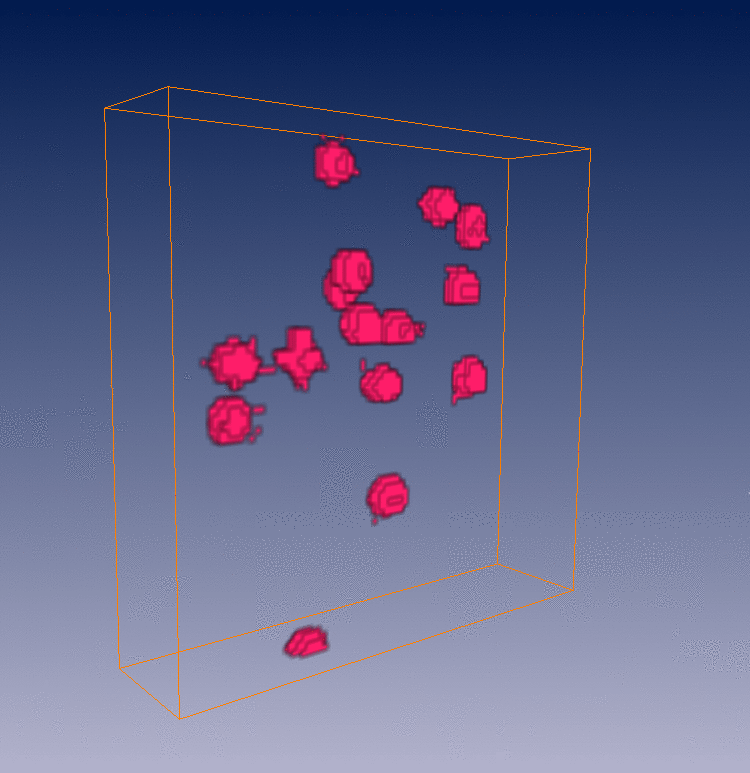
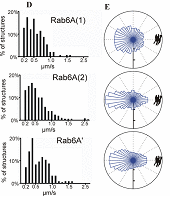
Analyse de trajectoires 4D des vésicules par la méthode des kymogrammes |
V. Racine, J. Salamero, V. Fraisier, A. Trubuil and J.B. Sibarita. Visualization and quantification of vesicle trafficking on a three-dimensional cytoskeleton network in living cells. J. Microscopy, 225(3): 213-227, 2007. Abstract. Recent progress in biology and microscopy has made it possible to acquire multidimensional data on rapid cellular activities. Unfortunately, the data analysis needed to describe the observed biological process still remains a major bottleneck. We here present a novel method of studying membrane trafficking by monitoring vesicular structures moving along a three-dimensional cytoskeleton network. It allows the dynamics of such structures to be qualitatively and quantitatively investigated. Our tracking method uses kymogram analysis to extract the consistent part of the temporal information and to allow the meaningful representation of vesicle dynamics. A fully automatic extension of this method, together with a statistical tool for dynamic comparisons, allows the precise analysis and comparison of overall speed distributions and directions. It can handle typical complex situations, such as a dense set of vesicles moving at various velocities, fusing and dissociating with each other or with other cell compartments. The overall approach has been characterized and validated on synthetic data. We chose the Rab6A protein, a GTPase involved in the regulation of intracellular membrane trafficking, as a molecular model. The application of our method to GFP-Rab6A stable cells acquired using fast four-dimensional deconvolution video-microscopy gives considerable cellular dynamic information unreachable using other techniques. |
Reconstruction de trajectoires partielles 4D des vésicules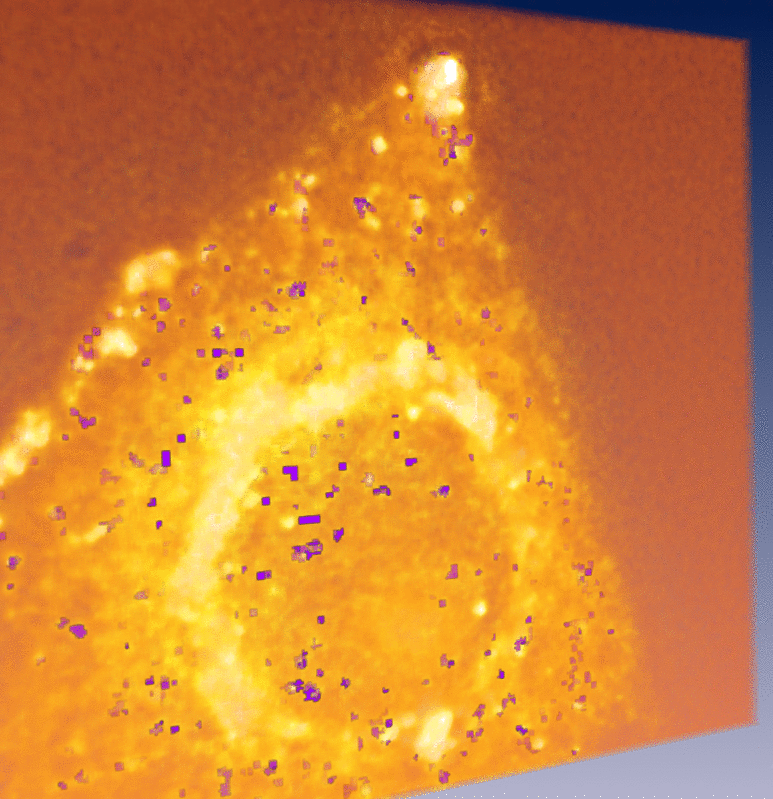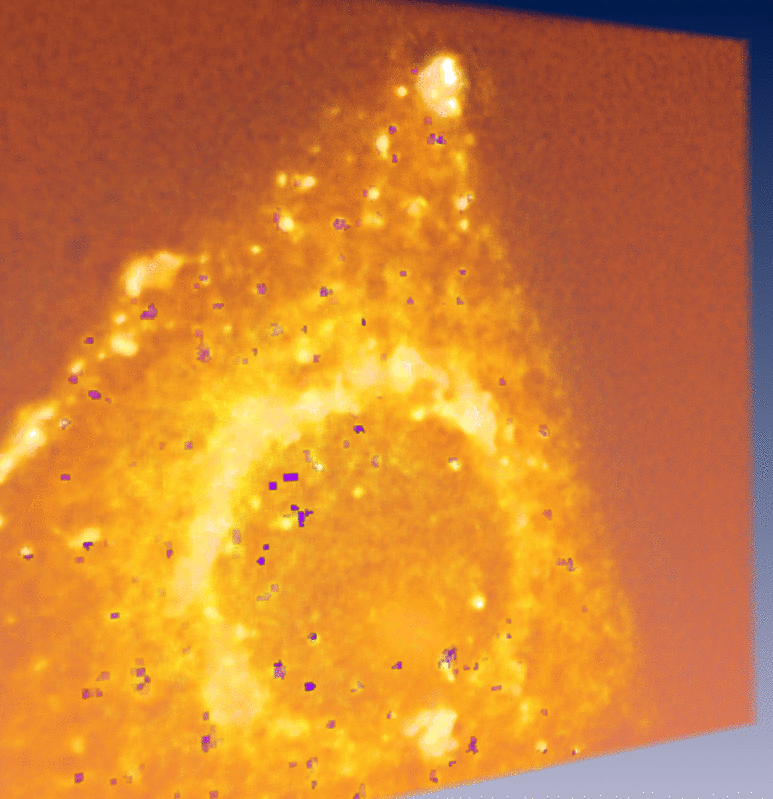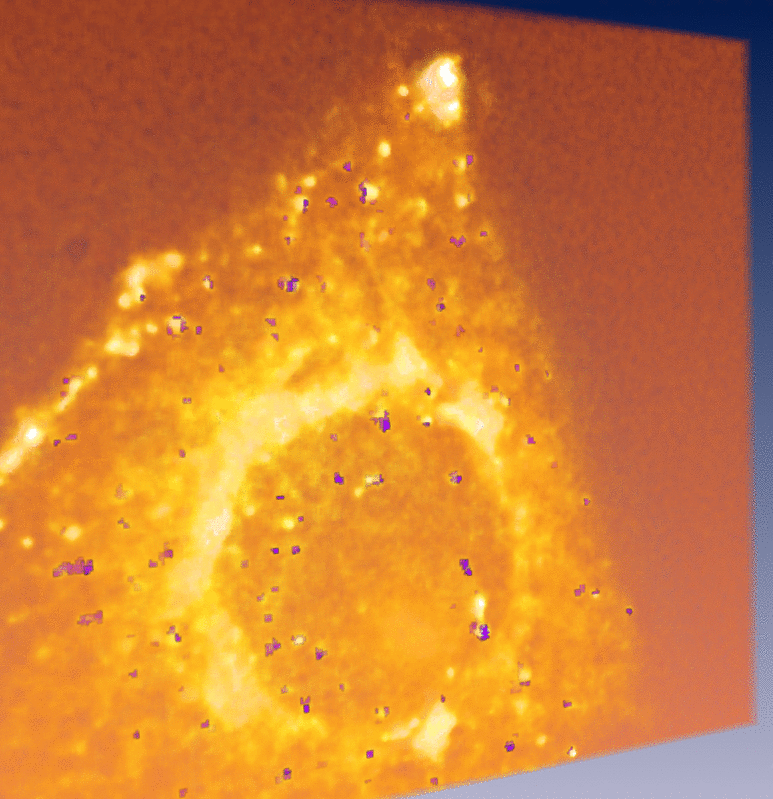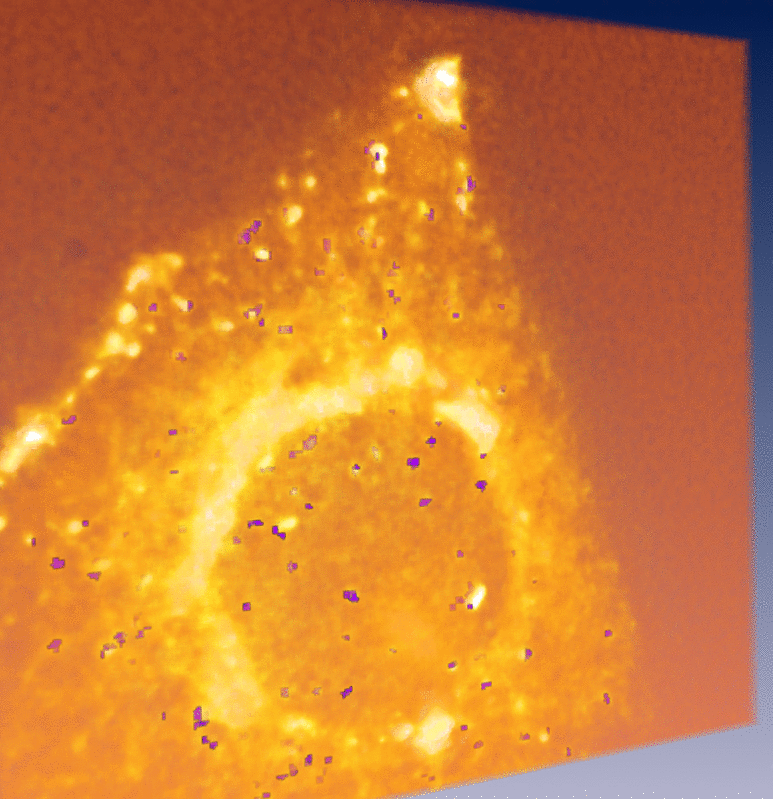 |
I. Bechar and A. Trubuil. Reliable motion detection and Analysis in live-cell imaging In Proc. Int. Symp. on Biomedical Imaging (ISBI'07) Arlington, USA, 2007. Abstract. We describe an alternative method to tracking for analyzing the motion of fluorescent spots in 3D+time video-microscopy. First, we realize motion detection by viewing the 3D+T spatio-temporal as a single 4D volume and detecting simultaneous significant change in the fluorescence of groups of neighboring voxels as spatio-temporal locations with high intensity. This step outputs spatio-temporal locations of motion and are next used to detect the moving spots and making statistical analysis of their motion. We show in particular that we are able to estimate the histogram of the motion speed using only the so segmented spatio-temporal locations. Simulated sequences are used to learn some of the parameters of this second step of our method. Results obtained on simulated sequences as well as on video-microscopic sequences are shown.
|
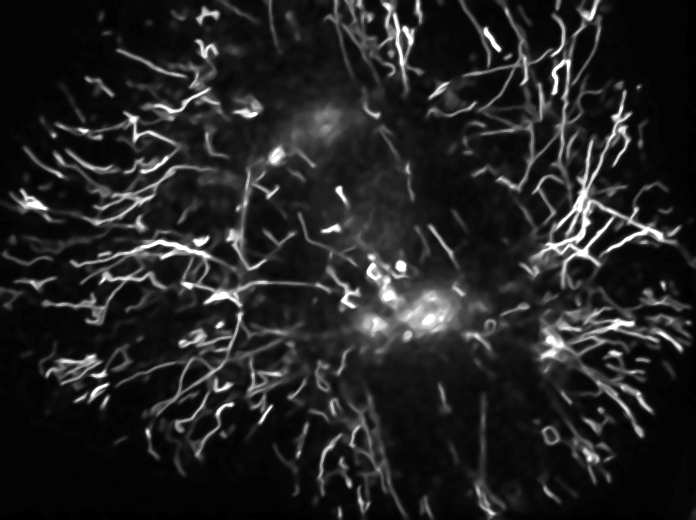
Suivi 4D de vésicules par une méthode "connexionniste" et analyse du module des vitesses
|
V. Racine. Quantification des dynamiques cellulaires par analyse de données de vidéo-microscopie 3D+t. Thèse de l'Université de Paris 6, décembre 2006. |
Simulation partielle du traffic de vésicules sur réseaux de microtubules - Analyse des parcours des particules en mouvement par graphes dynamiques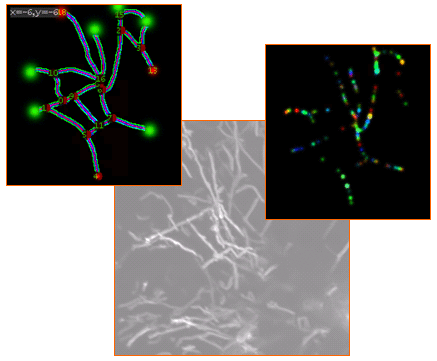 |
J. Boulanger, C. Kervrann, P. Bouthemy. Simulation and estimation of fluorescence in microscopy image sequence . In Proc. MICCAI'06 workshop on Microscopic Image Analysis and Applications to Biology (MIAAB'06) , Copenhagen, Denmark, 2006. Abstract. Image sequence analysis in video-microscopy for life sciences now has gained imp ortance since molecular biology is presently having a profound impact on the way research is being conducted in medicine. However, image processing techniques that are curre ntly used for modeling intracellular dynamics are still relatively crude. Indeed, com plex interactions between a large number of small moving particles in a complex scene cannot be easily modeled, which limits the performance of object detection and tracking algorithms. This motivates our present research effort which is to develop a general estimation/simulation framework able to produce image sequence s showing small moving spots in interaction and with variable velocities, corre sponding to intracellular dynamics and trafficking in biology. It is now well e stablished that spot trajectories can play a role in analysis of living cell dyn amics and simulate realistic image sequences is then of major importance. We dem onstrate the potential of the proposed simulation/estimation framework in expe riments, and show that this approach can be also used to evaluate the performan ce of object detection/tracking algorithms in video-microscopy.
|
Animation
- Réunion des partenaires du 21/12/04
- Début de thèse de Ikhlef Béchar (bourse ACI-IMPBIO) en janvier 2005
- Séjour de Jérome Boulanger (IRISA - Rennes) à l'INRA Jouy-en-Josas (du 03/02/05 au 04/02/05)
- Séjour de Victor Racine (Institut Curie - Paris) à l'IRISA-Rennes (du 28/02/05 au 03/03/05)
- Présentation du projet à la journée ACI-IMPBio (05/07/05)
- Visite de 2 jours de Ch. Kervrann et J. Boulanger à l'Institut Curie (28-29/09/05)
- Réunion des partenaires du 03/01/06
- Visite de 2 jours de Ch. Kervrann et J. Boulanger à l'Institut Curie (21-22/03/06)
- Réunion régulière tous les 2 mois à l'Intitut Curie au cours du seconde semestre 2006
- Réunion des partenaires du 24/01/07 à Rennes
- Journées Scientifiques et Médicales de l'Institut Curie (09/02/07)
- Visite de I. Béchar à l'Intitut Curie (03/07)
Publications
- C. Gueudry, C. Favard, V. Fraisier, V. Racine, J. Salamero, J.B. Sibarita. Fast 4D-FRAP imaging for analysing diffusion and membrane exchanges in living cells. Focus on Microscopy (FOM 2007), Valencia, Spain, 2007.
- J. Boulanger. Estimation non-paramétrique et contributions à l'analyse de séquences d'images. Modélisation, simulation et estimation du trafic intra-cellulaire dans des séquences de vidéo-microscopie. Thèse de l'Université de Rennes 1, Mention Traitement du Signal et des Télécommunications, janvier 2007.
- V. Racine. Quantification des dynamiques cellulaires par analyse de données de vidéo-microscopie 3D+t. Thèse de l'Université de Paris 6, décembre 2006.
- V. Racine, J. Salamero, V. Fraisier, A. Trubuil and J.B. Sibarita. Visualization and quantification of vesicle trafficking on a three-dimensional cytoskeleton network in living cells. J. Microscopy, 225(3): 213-227, 2007.
- I. Bechar and A. Trubuil. Reliable motion detection and Analysis in live-cell imaging In Proc. Int. Symp. on Biomedical Imaging (ISBI'07) Arlington, USA, 2007.
- S. Miserey-Lenkei, A. Couedel-Courteille, E. Del Nery, S. Bardin, M. Piel, V. Racine, J.B. Sibarita, F. Perez, M. Bornens and B. Goud. A role for the Rab6A' GTPase in the inactivation of the Mad2-spindle checkpoint. EMBO J. 25(2):278-89, 2006.
- I. Bechar and A. Trubuil. A bidimensional signal processing approach to vesicle trafficking analysis in 3D+T fluorescence videomicroscopy. In Proc. MICCAI ´06 - Workshop on Biophotonics Imaging for Diagnostics and Treatment, Copenhagen, Denmark, 2006.
- J. Boulanger, C. Kervrann, P. Bouthemy. Simulation and estimation of fluorescence in microscopy image sequence . In Proc. MICCAI'06 workshop on Microscopic Image Analysis and Applications to Biology (MIAAB'06) , Copenhagen, Denmark, 2006.
- V. Racine, A. Hertzog, J. Jouaneau, J. Salamero, C. Kervrann and J.B. Sibarita. Mutliple target tracking of 3D fluorescent objects based on simulated annealing. In Proc. Int. Symp. on Biomedical Imaging (ISBI'06), Arlington, USA, 2006.
- J. Boulanger, C. Kervrann, P. Bouthemy. Estimation of dynamic background for fluorescence video-microscopy . In Proc. Int. Conf. Image Processing (ICIP'06) , Atlanta, USA, Octobre 2006.
- J. Boulanger, C. Kervrann, P. Bouthemy. An adaptive statistical method for 4d-fluorescence image sequence denoising with spatio-temporal discontinuities preserving . In Biophotonics for Life Sciences and Medicine , F. Faugel, P. Smigielski, A. Brandenburg, J.Fontaine, Eds., Fontis Media SA, Lausanne, Switzerland, pp. 97-113, 2006.
- J.B. Sibarita, V. Racine and J. Salamero. Quantification of membrane trafficking on a 3D cytoskeleton network in living cells. . In Proc. Int. Symp. on Biomedical Imaging (ISBI'06), Arlington, USA, 2006.
- J. Langevin, M.J. Morgan, C. Rossé, V. Racine, J.B. Sibarita, S. Aresta, M. Murthy, T. Schwarz, J. Camonis and Y. Bellaïche/ Drosophila exocyst components Sec5, Sec6, and Sec15 regulate DE-Cadherin trafficking from recycling endosomes to the plasma membrane . Developmental Cell, 9:365-376, 2005.
- M. Thery, V. Racine, A. Pepin, M. Piel, Y. Chen, J.B. Sibarita, M. Bornens. The extracellular matrix guides the orientation of the cell division axis.Nature Cell Biology, 7(10):947-953, 2005.
- C. Angenieux, V. Fraisier, B. Maitre, V. Racine, N. van der Wel, D. Fricker, F. Proamer, M. Sachse, J.P. Cazenave, P. Peters, B. Goud, D. Hanau, J.B. Sibarita, J. Salamero and H. de la Salle. The cellular pathway of CD1e in immature and maturing dendritic cells. Traffic.6(4):286-302, 2005.
- C. Kervrann, J. Boulanger and P. Bouthemy. Spatio-temporal analysis in 4D video-microscopy . ERCIM News No.60, Special Theme: Biomedical Informatics, January 2005.
- J. Boulanger, C. Kervrann and P. Bouthemy. An adaptive statistical method for 4D-fluorescence image sequence denoising with spatio-temporal discontinuities preserving . In Imaging for Medical and Life Sciences (IMVIE2), Illkirch/Strasbourg, France, 2005.
- J. Boulanger, C. Kervrann and P. Bouthemy. An adaptive statistical method for denoising 4D fluorescence image sequences denoising with spatio-temporal discontinuities preserving . In Proc. Int. Conf. Image Processing (ICIP'05), Genova, Italy, 2005.
- J. Boulanger, C. Kervrann and P. Bouthemy. Adaptive spatio-temporal restoration for 4D fluoresence microscopic imaging. In Proc. Int. Conf. on Medical Image Computing and Computer Assisted Intervention (MICCAI'05), Palm Springs, USA, 2005.
- J. Boulanger, C. Kervrann and P. Bouthemy. Approche statistique adaptative pour le filtrage de sequences 3D+t d'images de microscopie de fluorescence . In Journées des Jeunes Chercheurs en Vision par Ordinateur (ORASIS'05), Clermont-Ferrand, France, 2005.